En bref :
La culture numérique devient une exigence pour chaque discipline, mais ce que cela peut signifier concrètement, et ce que cela pourrait apporter en classe n’a peut-être pas encore été suffisamment partagé et discuté avec les enseignants,…
Un projet: La biologie numérique : quelles opportunités pour mieux enseigner ? a élaboré et testé de nombreux scénarios.
On y trouve plus de 24 exemples en génétique, en évolution, en écologie, en physio, en immunologie, etc. ainsi que 10 autres proposés par le SIB.
Une formation continue PO 425 propose de les découvrir : Les opportunités de la biologie numérique : quelques applications en classe
Délai pour l’inscription : 28 septembre 2021
JTS présente plus bas un exemple : de solides preuves de l’évolution trouvées dans des données authentiques accessibles en classe. Ce scénario a été utilisé par plusieurs enseignants dans des classes de DF et OS. Ils ont relevé la focalisation sur la biologie : les aspects « ordinateur » n’ont guère posé de problèmes, et le TP a suscité de belles questions de la part des élèves.
L’évolution,… si difficile à enseigner !
Face à des idéologies, des religions, parfois des pressions dans l’école pour ne pas faire de vagues, il n’est guère facile de se positionner comme scientifique pour enseigner le modèle explicatif central de la biologie : « Rien n’a de sens en biologie si ce n’est à la lumière de l’évolution » Dobzhansky, T. (1973) Trad.C’est particulièrement pertinent en cette période ou un certain virus évolue devant nos yeux
Si l’enseignement de la biologie ne peut pas exiger la croyance (qui appartient au religieux, découle des valeurs et des appartenances), on peut demander aux élèves de savoir prédire ou expliquer en utilisant les explications scientifiques de l’évolution
JTS a développé cette approche ici : S’agit-il de croire en l’évolution ou savoir utiliser des modèles de l’évolution? Aider les élèves à trouver des preuves de l’évolution, ( sans attendre des millions d’année, puisqu’ils veulent avoir leur matu’ bientôt…:-) et pouvoir y appliquer ces modèles, composer et tester des hypothèses dans un TP est devenu possible.
Un projet avec la DGES II présente de nombreux exemples: La biologie numérique : quelles opportunités pour mieux enseigner ?
- Une formation continue est proposée pour les découvrir : PO 425
- Anat Yarden, chercheure présentera une étude Should we relate to students’ religious faith when teaching evolution? lors d’un Webinar (Zoom) de LS2 le 17 Novembre. Ouvert à tous sur inscription auprès de LS2
Un exemple
Ce serait bien si les élèves pouvaient vérifier que les espèces ont une origine commune dont elles divergent avec le temps et les conditions du milieu…
Au cœur – mais pas toujours explicité – des enseignements de l’évolution et de la phylogénie, il y a l’origine commune et la divergence au cours du temps et selon les conditions du milieu. On peut désormais aisément explorer des données authentiques pour observer cette unicité fondamentale du vivant (évidente pour le.la biologiste mais pas pour nos élèves cf. JTS ici et (Nehm, 2016)) que masque l’extraordinaire bio-diversité apparente ? Comment pourrait-on mettre en évidence, ou même faire découvrir par les élèves cette similitude fondamentale dans les fonctionnements d’un koala et d’une truite, d’une levure et d’un.e humain.e ?
Un TP pour aider les élèves à vérifier empiriquement l’évolution et ses mécanismes !
La biologie numérique offre désormais des opportunités pour aborder les questions de l’évolution de manière expérimentale. Un exemple : Comme de très nombreuses protéines sont présentes chez de multiples espèces animales et autres, on devrait pouvoir observer cette origine commune dans la similitude des séquences des protéines, non ?
Il est devenu aisé d’exploiter avec les élèves des données authentiques sur UniProt où des traces concrètes de l’origine commune de très nombreux organismes sont manifestes.
Constater que les séquences de l’insuline, de l’EPO, d’une Histone, de CFTR(exemple ici), etc. sont très similaires chez de nombreuses espèces, n’est-ce pas une belle manière de faire éprouver aux élèves cette unicité fondamentale?
L’insuline a des zones communes nombreuses mais varie dans bien des régions, alors que l’Histone (H4) est presque identique entre de très nombreux organismes.
![]() encourage le lecteur à trouver le protocole détaillé dans le projet d’origine : ici
encourage le lecteur à trouver le protocole détaillé dans le projet d’origine : ici 
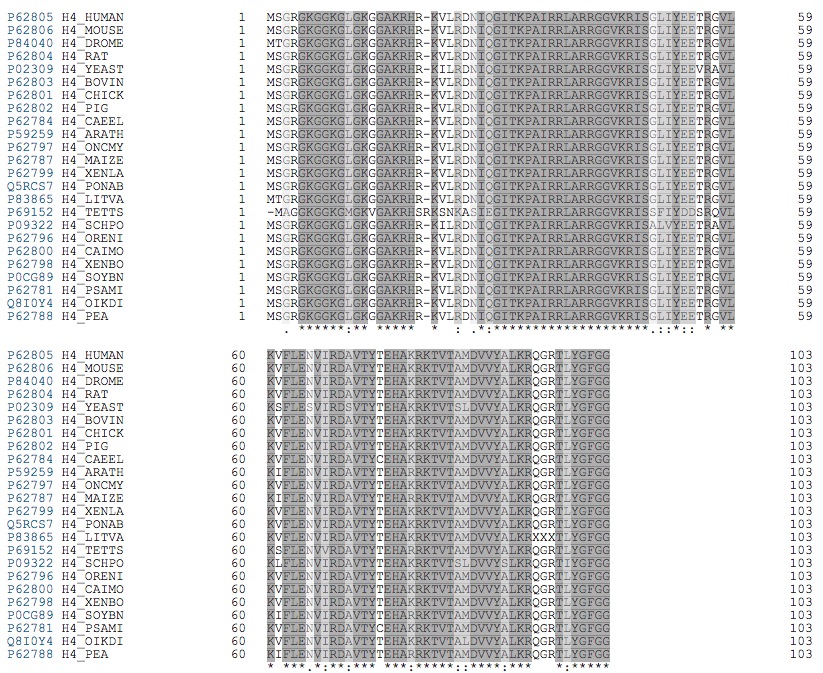 Fig 3: Gauche : alignement de l’insuline . Droite : alignement de l’Histone H4 de divers organismes (humain, bovin, levure, saumon, pois, etc). Notez les zones sombres (=séquence identique ou grisé (=similaires) (cliquer pour agrandir) [img]. Source :projet Bioinformatique : opportunités pour l’enseignement plus de détails ici
Fig 3: Gauche : alignement de l’insuline . Droite : alignement de l’Histone H4 de divers organismes (humain, bovin, levure, saumon, pois, etc). Notez les zones sombres (=séquence identique ou grisé (=similaires) (cliquer pour agrandir) [img]. Source :projet Bioinformatique : opportunités pour l’enseignement plus de détails ici
On peut par exemple attribuer à chaque groupe de 2 élèves une protéine différente et leur faire constater eux-mêmes que les plus similaires à l’humain sont chaque fois le chimpanzé ou le gorille, mais qu’ils sont assez différents du rat et la souris, qui sont tous deux très différents de la levure par exemple. ![]() encourage le lecteur à aller vérifier dans le projet d’origine : ici Les élèves pourront constater sur l’ensemble des écrans de la classe – même à distance- une grande zone sombre ( similitude) dans tous les cas : une très convaincante illustration de l’unicité du vivant
encourage le lecteur à aller vérifier dans le projet d’origine : ici Les élèves pourront constater sur l’ensemble des écrans de la classe – même à distance- une grande zone sombre ( similitude) dans tous les cas : une très convaincante illustration de l’unicité du vivant 
Fig 4: Lorsque les élèves réalisent un alignement -chacun pour une protéine différente – chez divers organismes les zones sombres reflétant la similitude sont visibles d’un coup d’oeil. Source :projet Bioinformatique : opportunités pour l’enseignement plus de détails ici
Pour constater la divergence, on peut ensuite leur demander de trouver les organismes les plus proches de l’humain, de la souris,…) pour chacune des protéines étudiées par la classe. Constater que les résultats sont convergents. On peut par exemple demander aux élèves d’essayer de trouver une autre explication que l’origine commune et l’évolution indépendante dans des milieux différents depuis la divergence.
![]() encourage le lecteur à aller vérifier dans le projet d’origine : ici UniProt produit un arbre des différences qui peut aider (mais n’est pas un vrai arbre phylogénétique plus de détails ici)
encourage le lecteur à aller vérifier dans le projet d’origine : ici UniProt produit un arbre des différences qui peut aider (mais n’est pas un vrai arbre phylogénétique plus de détails ici) 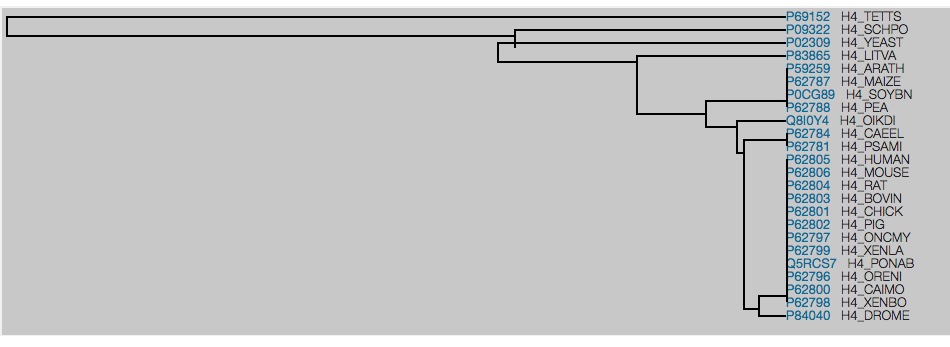
Fig 5: un arbre basé sur les différences observées entre les séquences, plus de détails ici) [img]. Source : projet Bioinformatique : opportunités pour l’enseignement Ce petit résumé donne peut-être envie d’aller voir de plus près ![]() encourage le lecteur à aller vérifier dans le projet d’origine : ici
encourage le lecteur à aller vérifier dans le projet d’origine : ici
23 autres exemples !
De nombreux autres exemples sont présentés ici ![]() La biologie numérique : quelles opportunités pour mieux enseigner ? Pour se apprivoiser et adapter ces exemples à ses classes et pratiques, une formation continue est proposée, avec Marie Claude Blatter du SIB et François Lombard de l’UniGE.
La biologie numérique : quelles opportunités pour mieux enseigner ? Pour se apprivoiser et adapter ces exemples à ses classes et pratiques, une formation continue est proposée, avec Marie Claude Blatter du SIB et François Lombard de l’UniGE.
COURS PO-425 – Les opportunités de la biologie numérique : quelques applications en classe
Délai pour l’inscription : en principe 28 septembre 2021
Objectifs
– Connaître plusieurs scénarios d’usage en classe, leurs insertions envisageables
– Débattre au sujet : des applications possibles en classe (comment et où inclure cette nouvelle biologie dans ses enseignements, en fonction des publics et des programmes… ), des limites de ces techniques et méthodes, des banques de données biologiques facilement accessibles en classe – Echanger à propos des différentes expériences des participant-e-s – Prendre la mesure de la place que la biologie numérique s’est attribuée dans la recherche en biologie- – Se faire une opinion sur les implications de tout ceci dans la manière d’aborder les programmes dans ses enseignements
– Démonstration de plus de 20 séquences/scénarios testés dans et applicables à des classes de biologie de l’enseignement secondaire (séquences combinables entre elles ou utilisables séparément, pouvant servir de base à une présentation interactive ou à un TP); ces séquences ont été choisies pour correspondre à des concepts difficiles pour les élèves ou à des sujets délicats à traiter – Présentation des thèmes suivants : biologie moléculaire(ADN, protéine), structure 3D des protéines (lien forme-fonction), introduction à l’évolution (preuves de l’évolution), maladie et médicaments, unicité et diversité du vivant (écologie – évolution) – Discussion des usages et partage d’expériences des participant-e-s
Date Mercredi 20 octobre 2021 De 13h30 à 17h00 : Formation en ligne
Intervenant(e)(s)
Dr Marie-Claude Blatter, institut suisse de bioinformatique (SIB)
Dr François Lombard, chargé de projet, Jump-To-Science et La biologie numérique : quelles opportunités pour mieux enseigner ?
https://outil.ge.ch/site/formation/rh/PO-425.html S’inscrire ![]() Depuis internet
Depuis internet
Références:
- Banta, L. M., Crespi, E. J., Nehm, R. H., Schwarz, J. A., Singer, S., Manduca, C. A., … Caporale, L. (2012). Integrating Genomics Research throughout the Undergraduate Curriculum: A Collection of Inquiry-Based Genomics Lab Modules. Cell Biology Education, 11(3), 203‑208. https://doi.org/10.1187/cbe.11-12-0105
- Coley, J. D., & Tanner, K. (2015). Relations between Intuitive Biological Thinking and Biological Misconceptions in Biology Majors and Nonmajors. CBE-Life Sciences Education, 14(1), ar8. https://doi.org/10.1187/cbe.14-06-0094
- Dobzhansky, T. (1973). Nothing in biology makes sense except in the light of evolution. American Biology Teacher, 35(3), 125‑129.
- Nehm, R. H. (2016). Keynote :Thinking about evolutionary change: concepts, contexts, and cognitive coherence. Paper presented at the ERIDOB 2016, Karlstad. Nehm ERIDOB print.pdf
- Perkel, J. M. (2017). How bioinformatics tools are bringing genetic analysis to the masses. Nature News, 543(7643), 137. https://doi.org/10.1038/543137a
