L’évolution : au coeur de la biologie mais contre-intuitive et si souvent communiquée de manière erronée
Des données pour vérifier, appliquer les explications biologiques accessibles en classe
Cette biologie numérique (bioinformatiques) est un des effets positif de la numérisation de la société (et de la science) dont chacun a pu voir les effets complexes, parfois positifs parfois négatifs dans l’enseignement à distance.
Jump-To-Science a sélectionné pour vous des données et des réflexions qui pourraient être utiles pour aider les élèves à comprendre l’évolution – et comment la sélection de certains variants pourraient influencer la propagation du virus du COVID dans un milieu que les mesures sanitaires ont changé.
La différenciation des souches – depuis décembre 2019 et à travers le monde à partir de Wuhan

Fig 1: Les différentes souches apparues au cours du temps et leur répartition en fonction du milieu [img]. Source :https://nextstrain.org/ncov/global
 encourage le lecteur à aller vérifier dans le site d’origine : ici
encourage le lecteur à aller vérifier dans le site d’origine : ici
Des données pour discuter l’évolution en classe
Il peut être intéressant d’explorer avec les élèves la puissance explicative des modèles de l’évolution vus en classe : Discuter si ces données authentiques pourraient s’expliquer (avec les modèles de sélection naturelle vus en classe) par la sélection des souches plus adaptées au milieu différent formé par les comportements qui changent (observation des règles de distanciation, port du masque, lavage des mains, etc.), ou la génétique, ou des autres facteurs (pyramide des âges ?, … ).
Les mutations du virus SARS-CoV-2
Le virus du COVID-19 évolue relativement lentement par rapport à celui du VIH (Callaway, E. 2020) – en effet une enzyme NSP14 répare la plupart des erreurs dans la copie de son ARN.
Le rôle, la séquence et la structure 3D de chacune des protéines du virus ici :
- Corum, J., & Zimmer, C. (2020). Bad News Wrapped in Protein : Inside the Coronavirus Genome. The New York Times, 3 avril. https://www.nytimes.com/interactive/2020/04/03/science/coronavirus-genome-bad-news-wrapped-in-protein.html | intranet.pdf
- Plus austère : les données de référence dans l’entrée GenBank correspondant au génome de référence du virus : https://www.ncbi.nlm.nih.gov/nuccore/NC_045512.2?report=genbank
Mutation-processus ou mutation-résultat?
Une difficulté pour les élèves résulte du fait que ce terme mutation désigne aussi bien les processus qui produisent un changement dans la séquence d’ADN ou d’ARN (erreurs de copie, substances mutagènes, rayonnements ionisants,…) que le résultat de ce changement : la séquence différente – plutôt appelé variant dans les banques de données (exemple) – et parfois allèle dans le modèle de Mendel.
« Le virus à muté pour devenir plus virulent » (cherchez les erreurs dans cette phrase)
SARS-CoV-2 évolue à mesure qu’il se propage et des mutations ont été observées explique Callaway, E. (2020). C’est sans parler des nombreuses qui se sont produites et n’ont pas pu être détectées car résultant en un virus non fonctionnel. Il y a donc de très nombreuses mutations dans tous les SARS-CoV-2 à travers le monde. Il faut noter qu’on parle de la fréquence des mutations observées et non de toutes les mutations qui se produisent plus ou moins au hasard dans toute la séquence
Cette disparition des mutations défavorables renforce une conception fréquente : que le virus ne muterait que dans le sens de devenir plus virulent (mieux adapté à son milieu…) . Et souvent on le décrit comme ayant des intentions mauvaises et engagé dans une guerre contre la science et la médecine. Ces explications finalistes font obstacle à une compréhension des mécanismes biologiques qui permet de prédire et expliquer ce qui se passe.
Des données sur LES mutations; disponibles en classe
Pour illustrer ou faire éprouver dans des activités pour les élèves ces affirmations, on dispose de données authentiques, actuelles et accessibles en classe.
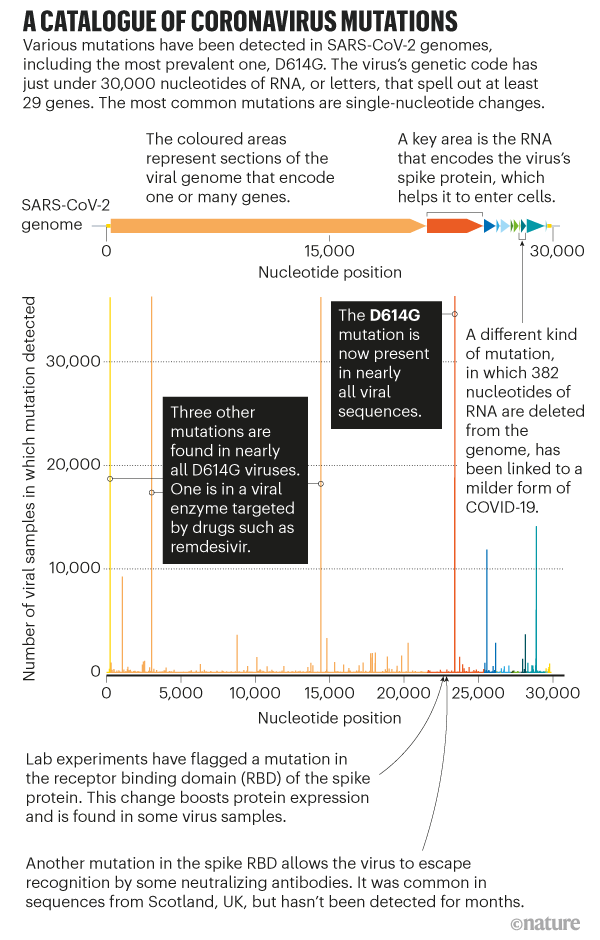
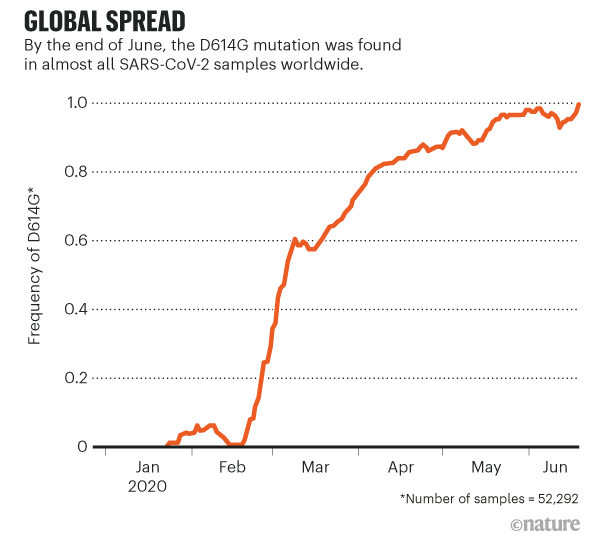
Fig 2: Fréquence des mutations observées en fonction de la position dans la séquence [img]. Source : Sources: L. Van Dorp et al. (http://go.nature.com/3GSRNH6); Refs 2, 11, 12; B. E. Young et al. Lancet 396, 603–611 (2020)
Korber, B., et al (2020) ont comparé les innombrables séquences du virus disponibles et ont vu la mutation D614G apparaître de plus en plus fréquemment dans des échantillons de personnes atteintes de COVID-19. À la position 614e de la protéine Spike, l’acide aminé aspartate (D, en sténographie biochimique) était régulièrement remplacé par la glycine (G) en raison d’un défaut de copie qui modifiait un seul nucléotide dans le code ARN de 29903 bases du virus. Les virologues ont nommé la mutation D614G. (Callaway, E. 2020)Traduction
Une mutation change un peu la conformation de la protéine Spike par laquelle le virus s’accroche à nos cellules
Cette protéine Spike est celle par laquelle le virus (cf. Jump-To-Science 29 Avril 2020) se fixe sur ACE2 à la surface de nos cellules.
Quel est le rôle normal de la protéine ACE2 ?
Les élèves ( ou leur enseignant.e) peuvent trouver des éléments de réponse ici :
Où la protéine ACE2 est-elle exprimée dans le corps humain :
Figure 5 : Degré d’expression pour ACE2 trouvé dans proteinatlas.org
Une mutation SARS-Cov2 comme exemple d’évolution en marche
Fig 3: cliquer pour voir les différentes conformations «fermées» et «ouvertes» de la protéine Spike du virus SARS-CoV-2. Cette protéine se lie aux récepteurs des cellules humaines et permet l’entrée du virus. Une mutation fréquente (D614G cercle) semble favoriser les conformations ouvertes de la protéine, ce qui pourrait signifier que le virus peut pénétrer plus facilement dans les cellules. [img]. Source Callaway, E. (2020). Structural data from K. Shen & J. Luban
Une mutation qui donne aux virus qui la portent une plus grande infectiosité ?
Hou, Y. J. et al. (2020) ont analysé les effets de cette variante du gène Spike avec la mutation D614G. Elle semble favoriser les conformations ouvertes de la protéine Spike, et favoriserait la pénétration du virus peut pénétrer dans les cellules explique Callaway, E. (2020).
Fig 4: les variantes plus ouvertes de spike sont associées à une probabilité d‘infection plus grande [img]. Source : Callaway, E. (2020).
Voir et imprimer les formes différentes de la protéine Spike avec les élèves ?
- La protéine Spike @ UniProt : P0DTC2
- La structure 3D de la protéine Spike @PDB visualiser 6VXX (fermée 6VXX ) et 6VSB (partiellement ouverte) la convertir pour imprimante 3D
La saturation du milieu augmente la sélection
Stephen J. Gould (1977) résumait le mécanisme de l’évolution en 2 faits et une conclusion inévitable (ici)
- 1. Les organismes varient au hasard : ces variations sont (au moins en partie), transmises à leur descendance.
- 2. Les organismes produisent plus de descendants qu’il n’en pourra survivre cet endroit
- 3 . En moyenne les descendants dont les variations sont favorisées par l’environnement survivent et prolifèrent. Les variations favorables s’accumulent par conséquent dans les populations par sélection naturelle.
Gould, S. J. (1977)Traduction personnelle
Il est rare d’observer la situation inverse, où un nouvel organisme apparait dans un milieu qui ne le limite pas et où il n’y a que peu de sélection. On pourrait voir un parallèle avec l’explosion radiative vers le début du Cambrien où (Gould, et al, 1993) dans le magnifique livre de la vie expliquent que la concentration d’Oxygène a rendu possible des parties dures et une diversité incroyable de formes d’organismes s’est multipliée presque sans concurrence pour un temps.
Une formation continue :
(Les membres Jump-To-Science peuvent obtenir ces articles…).
Références:
- Callaway, E. (2020). The coronavirus is mutating—Does it matter? Nature, 585(7824), 174‑177. https://doi.org/10.1038/d41586-020-02544-6
- CIIP. (2011). Plan d’études Romand (Mathématiques et sciences de la nature). Conférence intercantonale de l’instruction publique de la Suisse Romande et du Tessin,.
- Coley, J. D., & Tanner, K. (2015). Relations between Intuitive Biological Thinking and Biological Misconceptions in Biology Majors and Nonmajors. CBE-Life Sciences Education, 14(1), ar8. https://doi.org/10.1187/cbe.14-06-0094
- Corum, J., & Zimmer, C. (2020, avril 3). Bad News Wrapped in Protein : Inside the Coronavirus Genome. The New York Times. https://www.nytimes.com/interactive/2020/04/03/science/coronavirus-genome-bad-news-wrapped-in-protein.html | intranet.pdf
- Dobzhansky, T. (1973). Nothing in biology makes sense except in the light of evolution. American Biology Teacher, 35(3), 125‑129.
- Gould, S. J. (1977). Ever since Darwin: Norton New York.
-
Gould, S. J. et al. (1993). Le livre de la vie. Seuil
- Hou, Y. J. et al. (2020), SARS-CoV-2 D614G Variant Exhibits Enhanced Replication ex vivo and Earlier Transmission in vivo | bioRxiv (preprint). https://doi.org/10.1101/2020.09.28.317685
- Korber, B., Fischer, W., Gnanakaran, S., Yoon, H., Theiler, J., Abfalterer, W., Foley, B., Giorgi, E., Bhattacharya, T., Parker, M., Partridge, D., Evans, C., Freeman, T., de Silva, T., on behalf of the Sheffield COVID-19 Genomics Group, LaBranche, C., & Montefiori, D. (2020, avril 30). Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2. https://doi.org/10.1101/2020.04.29.069054
- Potvin, P. (2019). Faire apprendre les sciences et la technologie à l’école : Épistémologie, didactique, sciences cognitives et neurosciences au service de l’enseignant. Presses de l’université Laval. Extraits intranet.pdf
- Potvin, P., Sauriol, É., & Riopel, M. (2015). Experimental evidence of the superiority of the prevalence model of conceptual change over the classical models and repetition. J Res Sci Teach, 52(8), 1082‑1108. https://doi.org/10.1002/tea.21235
- Young, B. E., Fong, S.-W., Chan, Y.-H., Mak, T.-M., Ang, L. W., Anderson, D. E., Lee, C. Y.-P., Amrun, S. N., Lee, B., Goh, Y. S., Su, Y. C. F., Wei, W. E., Kalimuddin, S., Chai, L. Y. A., Pada, S., Tan, S. Y., Sun, L., Parthasarathy, P., Chen, Y. Y. C., … Ng, L. F. P. (2020). Effects of a major deletion in the SARS-CoV-2 genome on the severity of infection and the inflammatory response : An observational cohort study. The Lancet, 396(10251), 603‑611. https://doi.org/10.1016/S0140-6736(20)31757-8