Abstract
L’annonce du prix Nobel de chimie partagé avec un chercheur romand au style de communication assez chaleureuse et humaine donne un bon exemple pour dépasser le cliché du savant dans sa tour d’ivoire ou carrément dérangé. Ensemble ils ont mis au point une technique qui permet de figer et de déterminer la structure 3D des bio-molécules avec une précision grandement accrue.
« The Nobel Prize in Chemistry 2017 was awarded to Jacques Dubochet, Joachim Frank and Richard Henderson « for developing cryo-electron microscopy for the high-resolution structure determination of biomolecules in solution« . »
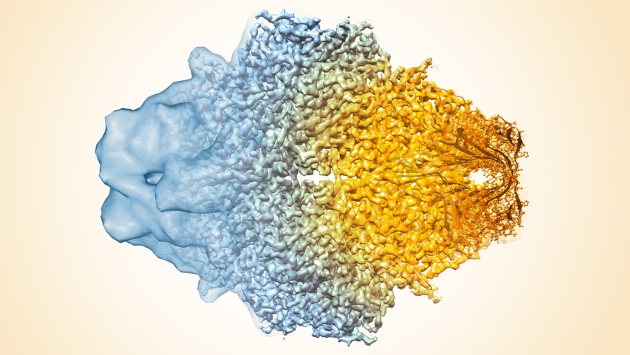
Fig 1: La Cryo-electron microscopy de proteins (ici l’enzyme β-galaxtosidase) a permis de passer de la carte de densité à basse résolution (à gauche) aux coordonnées atomiques à droite. [img]. Source :Cressey, D., & Callaway, E. (2017)
Jacques Dubochet raconte avec ironie que sa contribution a d’abord été rejetée par Nature et que cette technique a permis de très nombreuses découvertes abondamment publiées dans cette même revue… et fonde -avec deux autres – l’attribution du prix Nobel de chimie cette année.
A l’UniGE des chercheurs utilisent cette technique couramment. Dr Didier Perret du Chimiscope qui a travaillé avec Dubochet commente la technique avec le regard du connaisseur, et Prof. Paul Guichard à l’UniGE présente deux publications récentes dans Nature et montre comment la Cryo-EM a permis de nouvelles percées pour comprendre la maladie d’Alzheimer.
D’innombrables structures 3D produites par la recherche sont dans des banques de données librement accessibles – aussi en classe. Une formation continue offerte aux enseignants présentera les possibilités d’usage en classe de ces biomolécules à l’heure ou les imprimantes 3D deviennent courantes FC PO ici.
La microscopie électronique et la congélation instantanée de bio-molécules en solution pour déterminer leur structure 3-D
Comme le décrit Jacques Dubochet en 2012, (ici), à cette époque, sa découverte était si révolutionnaire que la revue NATURE, un des journaux en science les plus en vue, refusa son article. Les relecteurs ont justifié en indiquant : « you cannot bend nature« , qui signifiait que sa découverte allait au delà des lois physiques et que l’eau vitreuse dans un microscope était impossible. Plus de trente années plus tard, Jacques Dubochet reçoit le prix Nobel et NATURE publie aujourd’hui plusieurs dizaines d’articles par an utilisant cette méthode : la Cryo-microscopie (cryo-electron microscopy ou cryo-EM) qui permet de déterminer la structure 3-D de molécules (notamment protéines et acides nucléiques) dans leur état natif et à haute-résolution.
Notons que Dubochet n’est pas seul, une fois sur deux la publication fondatrice qui a valu le prix Nobel avait été refusée par Nature et Science. Il faut reconnaitre que ce doit être bien difficile de distinguer l’étrange qui est révolutionnaire de la masse d’articles simplement farfelus… Même les meilleurs éditeurs avec de bons reviewers n’y parviennent pas à coup sûr (cf. par exemple cet Editorial de Nature (2003), ici , ou ScienceAlert).
« La cryo-EM consiste à congeler très rapidement des échantillons biologiques purifiés dans un bain d’éthane liquide refroidit à la température de l’azote liquide (-196°C). Cette étape s’appelle la vitrification où les molécules d’eau n’ont pas le temps de prendre un arrangement cristallin comme lors de la formation de glace. Dans ces cas, les molécules d’eau gardent la même organisation qu’à l’état liquide tout en étant congelés. L’échantillon d’eau vitreuse qui contient les bio-molécules est ensuite transféré dans un microscope électronique afin de visualiser la structure des biomolécules à étudier.
Avant cette découverte, les bio-molécules étaient visualisées en enlevant l’eau autour des protéines, ce qui les déformait. Notre chercheur romand, Jacques Dubochet, a inventé cette méthode de vitrification (flash-freeze) qui permet de garder les échantillons dans leur état dit natif. Aujourd’hui cette méthode est utilisée à travers le monde pour résoudre à l’échelle atomique la structure des bio-molécules cellulaires afin de mieux comprendre leur fonctionnement. » Prof. Paul Guichard
Dr. Didier Perret du Chimiscope qui a travaillé avec les microscopes de Dubochet résume la technique et commente pour Expériment@l-Tremplins
L’invention de la microscopie électronique à transmission (TEM) en 1933 par Ernst Ruzka n’a été récompensée du Prix Nobel de physique qu’en 1986, conjointement à Heinrich Rohrer et Gerd Binnig pour leurs travaux chez IBM-Zürich ayant conduit à la création en 1981 du microscope à effet tunnel. C’est dire qu’il aura fallu 53 années pour que l’un des outils privilégiés des scientifiques passe du statut de simple instrument de visualisation à celui de découverte fondamentale permettant d’ouvrir des champs de compréhension des mécanismes atomiques et moléculaires à l’échelle nanoscopique !
Le Prix Nobel de chimie 2017 ne récompenserait-il alors qu’une « simple amélioration technique » de la microscopie ? Manifestement non ! Pour mesurer l’extraordinaire avancée des origines de la TEM à aujourd’hui, il faut comprendre que l’eau (qui constitue l’essentiel de la matière étudiée en biologie) et le haut vide (indispensable au cheminement des électrons dans la colonne du TEM) ne font simplement pas bon ménage.
Pour étudier un échantillon essentiellement aqueux, l’approche « traditionnelle » consiste à procéder à de fastidieuses étapes d’élimination de l’eau et d’inclusion en résine hydrophobe ; cette méthode normative est encore largement utilisée aujourd’hui malgré ses nombreux artefacts avérés, car elle produit des résultats qui sont aisément comparables dans le temps et d’un chercheur à l’autre.
Alternativement et pour contourner les dommages provoqués par la déshydratation, l’échantillon peut être inclus en résine hydrophile ; cette méthode, inventée en 1983, n’a jamais reçu les faveurs des biologistes malgré ses avantages documentés, car elle pose le problème de la comparaison de résultats obtenus dans des conditions différentes ; elle fait cependant fureur auprès des chimistes qui étudient les colloïdes et particules en milieu aquatique naturel.
Finalement et logiquement, la panacée consiste à conserver l’eau dans l’échantillon, en la refroidissant tellement rapidement qu’elle « vitrifie » (littéralement : formation d’une phase amorphe excluant la création de volumineux cristaux de glace fortement dommageables pour les fines et fragiles structures à étudier). Et étrangement, c’est avec de l’éthane liquide (-183 °C) ou du propane liquide (-188 °C), et non avec de l’azote liquide plus froid (-196 °C), que la congélation est la plus rapide et qu’elle permet de figer l’échantillon intact dans son milieu natif pour qu’il révèle sous le microscope toutes ses subtilités morphologiques sans modification. Cette découverte fondamentale vaut donc bien un Prix Nobel car elle révolutionne aujourd’hui la compréhension des systèmes biologiques.
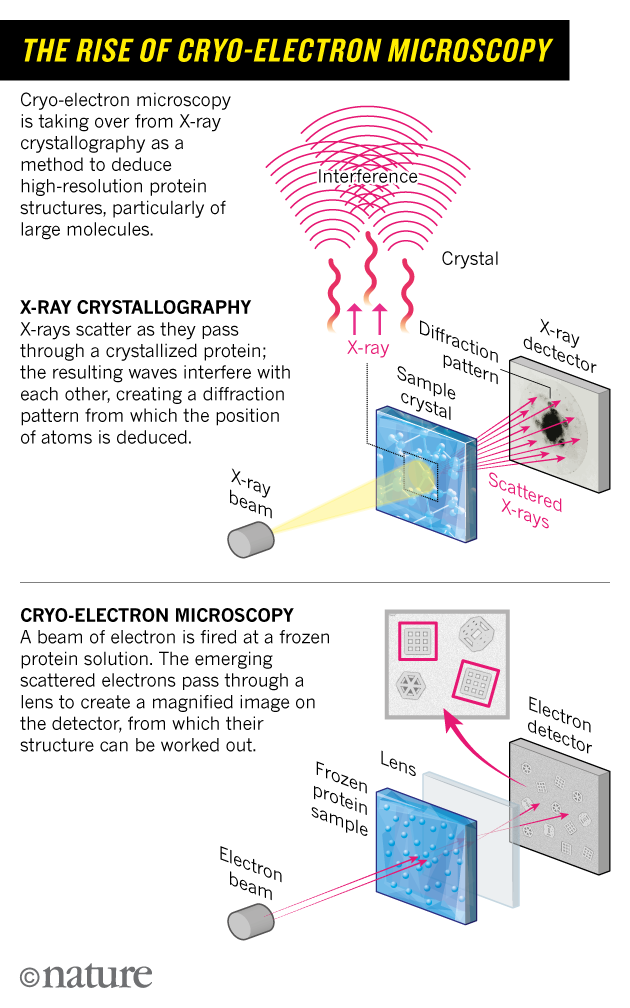
Fig 2: Les mécanismes de la cryo-microscopie – comparée à la cristallographie a rayons X [img]. Source :Cressey, D., & Callaway, E. (2017)
Pour plus de détails un review sur la technique :
- Dubochet, J. (2012). Cryo-EM—the first thirty years. Journal of Microscopy, 245(3), 221‑224. https://doi.org/10.1111/j.1365-2818.2011.03569.x
Un chercheur romand très humain
Peut-être que cette image moins austère peut aider certain-e-s élèves à envisager des études en science ?

Fig 3: Jacques Dubochet video Darius Rochebin (Emission Pardonnez-moi, ) [img]. Source : TSR
Jacques Dubochet video Darius Rochebin (Emission Pardonnez-moi, ) reçoit le Prix Nobel de chimie 2017
L’information depuis le site Nobel pour la chimie 2017
Read more about this year’s prize:
Des chercheurs à l’UniGE viennent de faire une découverte importante en utilisant la cryo-microscopie

Fig 4: La structure des TOROIDS révélée par cryoEM en blanc avec les structures atomiques des différentes protéines qui la composent en couleur. [img]. Source : Prouteau, et al. (2017)
Prof. Paul Guichard, un des coauteurs, précise : « Cette technique est installée à l’UniGE depuis plus de deux ans afin d’étudier des complexes macromoléculaires. En utilisant cet outil, Manoel Prouteau, collaborateur scientifique dans le laboratoire du Professeur Robbie Loewith à l’UniGE a pu révéler la structure d’un nouvel assemblage cellulaire, les TOROIDS.
Ces chercheurs ont découvert que l’enzyme TOR (Target Of Rapamycine), impliquée dans le développement et le vieillissement cellulaire, s’inactive en formant un complexe macromoléculaire lorsque la cellule est privée de glucose. Ces mêmes chercheurs ont également montré que cet assemblage peut se désassembler lorsque que le glucose est à nouveau présent et ainsi ré-activer la fonction de l’enzyme TOR. En utilisant la cryo-microscopie, le groupe du Professeur Loewith a découvert que cet assemblage était en fait une gigantesque structure en forme d’hélice. En formant cette hélice, les zones critiques à la fonction de TOR sont masquées, expliquant ainsi le mécanisme d’inactivation de l’enzyme. Cette découverte a été publié récemment dans le journal Nature: »
- Prouteau, M., Desfosses, A., Sieben, C., Bourgoint, C., Lydia Mozaffari, N., Demurtas, D., … Loewith, R. (2017). TORC1 organized in inhibited domains (TOROIDs) regulate TORC1 activity. Nature, 550(7675), 265‑269. https://doi.org/10.1038/nature24021
Une recherche sur les fibres amyloïdes fait la une de Nature
Prof. Paul Guichard précise l’importance cruciale qu’a pris la cryo-EM : « Pourquoi utiliser la cryo-microcopie pour étudier nos molécules ? Notre corps est fait de cellules qui sont elles mêmes composes de centaines de milliers de molécules. Ces molécules, qui ne font que quelques nanomètre de long ont des structures caractéristiques à leur fonction. Si la structure est altérée, sa fonction peut l’être aussi et entrainer des maladies. C’est le cas dans la maladie d’Alzheimer où la protéine Tau change de structure pour former des fibres amyloïdes, responsable du développement de la maladie. Grace à la cryo-microscopie, des chercheurs du laboratoire de biologie moléculaire à Cambridge (Fitzpatrick, A. W. P., et al., 2017) ont pu résoudre la structure des fibres amyloïdes il y a quelques mois et ainsi mieux comprendre comment ces fibres se forment. »
- Fitzpatrick, A. W. P., Falcon, B., He, S., Murzin, A. G., Murshudov, G., Garringer, H. J., … Scheres, S. H. W. (2017). Cryo-EM structures of tau filaments from Alzheimer’s disease. Nature, 547(7662), 185‑190. https://doi.org/10.1038/nature23002
Couverture du journal NATURE pour cette découverte:

Fig 5 : La structure des fibres amyloïdes résolue par cryoEM [img]. Source : Fitzpatrick, A. W. P., et al., 2017
Des protéines et d’autres biomolécules disponibles librement en 3D – même en classe
Une banque de données donne accès aux structures 3D obtenues par cryo-EM 
Sur la banque de données 3D RCSB PDB on trouve de très nombreuses molécules établies par Cryo-EM : par exemple le virus Zika
https://www.rcsb.org/pdb/ngl/ngl.do?pdbid=5IRE&bionumber=1
Comment exploiter ces structures 3D en classe ?
Disposer de ces magnifiques ressources ne suffit pas à ce que les apprentissages s’ensuivent automatiquement. Il y a toute une pédagogie à inventer.

Fig 7: L’usage en classe de modèles imprimés en 3D sera discuté dans une formation continue . Photo F. Lombard avec l’accord de l’élève.
L’usage en classe des structures 3D et leur matérialisation à l’aide d’imprimantes 3-D vous est offerte :
Une formation continue sur les protéines en relief et en volume est proposée le mercredi 21 mars 2018 après-midi, avec M.-C. Blatter, expert en banques de données (SIB), Daniel Schneider et Stéphane Morand, experts en impression 3D (TECFA) et François Lombard, (Collège Calvin, IUFE, TECFA).
Au cours de cette après-midi vous apprendrez à sélectionner une protéine ou un ARN, à trouver sa structure 3D dans une banque de données (souvent produite par Cryo-EM), à la visualiser à l’écran, à la convertir en format pour impression 3D. Vous verrez le processus d’impression d’une ou deux vous recevrez une structure 3D à emporter. Vous emporterez votre sélection de molécule 3D sur une clé USB pour aller l’imprimer.
Inscrivez-vous ici : PO-422 – La protéine en volume et en relief mercredi 21 mars 2018 après-midi
Références : (Les membres Expériment@l-Tremplins peuvent obtenir ces articles…).
-
Cressey, D., & Callaway, E. (2017). Cryo-electron microscopy wins chemistry Nobel. Nature, 550(7675), 167‑167. https://doi.org/10.1038/nature.2017.22738
- Dubochet, J. (2007). The Physics of Rapid Cooling and Its Implications for Cryoimmobilization of Cells. In Methods in Cell Biology (Vol. 79, p. 7‑21). Academic Press. https://doi.org/10.1016/S0091-679X(06)79001-X
- Dubochet, J. (2012). Cryo-EM—the first thirty years. Journal of Microscopy, 245(3), 221‑224. https://doi.org/10.1111/j.1365-2818.2011.03569.x
- Editorial, Nature (2003). Coping with peer rejection. Nature, 425(6959), 645‑645. https://doi.org/10.1038/425645a
- Fitzpatrick, A. W. P., Falcon, B., He, S., Murzin, A. G., Murshudov, G., Garringer, H. J., … Scheres, S. H. W. (2017). Cryo-EM structures of tau filaments from Alzheimer’s disease. Nature, 547(7662), 185‑190. https://doi.org/10.1038/nature23002
- Perret D., Leppard G.G., Mavrocordatos D. (2005). Microscopy applications – environmental. In P. Worsfold, A. Townshend, C. Poole (Eds), Encyclopedia of Analytical Science, 2nd edition, Vol. 6. p.65-73. Elsevier Academic, San Diego.
- Mavrocordatos D., Perret D., Leppard G.G. (2007). Strategies and advances in the caracterisation of environmental colloids by electron microscopy. In K.J. Wilkinson, J.R. Lead (Eds), Environmental Colloids and Particles – Behaviour, Separation and Characterisation. p. 345-404. IUPAC Series on Analytical and Physical Chemistry of Environmental Systems. Wiley, Chichester.
- Prouteau, M., Desfosses, A., Sieben, C., Bourgoint, C., Lydia Mozaffari, N., Demurtas, D., … Loewith, R. (2017). TORC1 organized in inhibited domains (TOROIDs) regulate TORC1 activity. Nature, 550(7675), 265‑269. https://doi.org/10.1038/nature24021


