La biologie; une discipline sans chiffres ?
Souvent les élèves comme les enseignants hésitent à aborder les aspects quantitatifs de la biologie- notamment faute de trouver facilement des chiffres pour préciser « souvent » « beaucoup » « minuscule ». Une base de données BioNumbers en rassemble une collection remarquable, et un site – Caladis.org – permet d’expliciter et de travailler dynamiquement sur les approximations et les degrés d’erreur. Par souci citoyen de former des adultes qui ne baissent pas les yeux pudiquement au premier chiffre et parce que la biologie est devenue numérique, certains proposent de développer les compétences bio-numériques.
En un clin d’oeil ? « M’sieur ça dure combien de temps un clin d’oeil ? »
On trouve dans cette base, (référencée Milo et al. (2010) Nucl. Acids Res. BNID 100706) BioNumbers sous le n° 100706 Le chiffre suivant
Average duration of a single eye blink | Homo sapiens | 0.1-0.4 | sec | 100706 | http://faculty.washi…
Selon cette source, un clin d’oeil dure donc 1 à 4 dixièmes de seconde.
La vitesse de réplication de l’ADN ?
La vitesse à laquelle avance la réplication est une question que les élèves posent parfois. La nécessité de nombreux « yeux de réplication » est logique si on a une idée de la longueur d’un chromosome et de la vitesse de réplication. (On peut calculer avec les élèves que si on a 3millards de bases par cellule, un chromosome fait environ (si je ne me trompe pas, j’ai un peu de dyslexie) 140 millions de bases (bien plus pour le 1 et bien moins pour le 22). A 33bases par seconde il faudrait de l’ordre de 3 millions de secondes ou un bon mois (env 36jours) pour le dupliquer ! Clairement ça ne peut pas suffire.
N.B: si vous trouvez une erreur ou une proposition d’amélioration, un complément c’est volontiers ! Vous êtes peut-être bien plus compétent que moi. N’hésitez pas a commenter le Blog Bio-Tremplins-Experiment@l : Ces réflexions sont destinées à susciter une réflexion et le désaccord me plait bien !
Rate of DNA replication | Human Homo sapiens | ~33 | nucleotides/sec | 104136 | Fangman WL, Brewer BJ….
Milo et al. (2010) Nucl. Acids Res. BNID 104136. BioNumbers
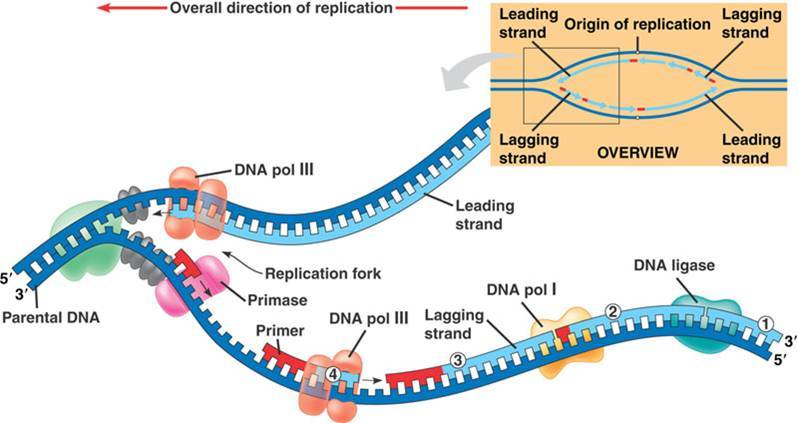
Fig 1: la vitesse à laquelle avance la réplication est donne du sens aux yeux de réplication. [img] source Campbell 2007
La réplication progresse à la vitesse de 33 nucléotides par seconde chez l’humain
Une très belle animation du Howard Hugues Medical Institute (HHMI) ici  permet de donner une idée de la vitesse de cette réaction (probablement pas à montrer au début du chapitre … si on veut pas effrayer les élèves ?) Intranet.mov si jamais vos écoles n’ont pas accès à Youtube ici
permet de donner une idée de la vitesse de cette réaction (probablement pas à montrer au début du chapitre … si on veut pas effrayer les élèves ?) Intranet.mov si jamais vos écoles n’ont pas accès à Youtube ici
Et chez d’autres organismes ?
DNA replication speed in vivo | Bacteria Escherichia coli | ~600 | bp/sec | 109251 | Reyes-Lamothe R, Possoz C…
Elle est donc de 600 bases à la seconde chez E. coli selon Reyes-Lamothe in Milo et al. (2010) Nucl. Acids Res. BNID 109251.
Il vaudrait la peine d’aller consulter cette source car la vitesse de ce processus varie surement avec la température… Probablement à 37°C ?
BioNumbers vous offre des détails ici Expériment@l-Tremplins vous offre ce tremplin vers les savoirs…
;-))
La distance entre deux origines de réplication de l’ADN ?
Le nombre d’yeux de réplication par chromosome peut être estimé si on a la distance entre deux origines de réplication.
Spacing between origins of DNA replication | Human Homo sapiens | ~100 | kb | 104137 | Fangman WL, Brewer BJ….
Milo et al. (2010) Nucl. Acids Res. BNID 104137 BioNumbers
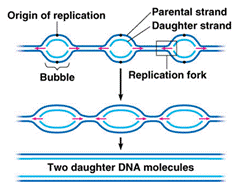
Fig 2: De nombreuses origines de réplication produisent plusieurs yeux de réplication. [img] source Campbell 2007
Selon Fangman, l’espace entre deux origines de réplications est d’environ 100’000 bases chez H. Sapiens
On peut donc reprendre notre chromosome moyen de 140 millions de bases et estimer (si je ne me trompe pas) qu’il y a 140’000’000/100’000 = 1’400 yeux de réplication pour un chromosome ( peut-être 2-3 k pour le chr 1 et sans doute moins de 500 pour le 22). Et chaque oeil mettrait environ une demi-heure à rejoindre le suivant.
Le taux de mutations par division ?
| Mutation rate per genome per replication | Human Homo sapiens | 0.16 (Table – link) | mutation/genome/replication | 100417 |
|
||
| Mutation rate per base pair per replication | Nematode Caenorhabditis elegans | 2.3E-10 (Table – link) | mutation/bp/replication | 100287 |
|
||
| Mutation rate per genome per replication | Bacteriophage Lambda | 0.0038 (Table – link) | mutation/genome/replication | 100222 |
|
||
| Mutation rate per base pair per replication | Human Homo sapiens | 5E-11 (Table – link) | Mutation/bp/replication | 105095 |
|
Milo et al. (2010) Nucl. Acids Res. BNID 100417, 100287, 100222, 105095. BioNumbers
Ainsi il y aurait une mutations par génome toutes les 6 réplication environ chez l’homme, ou 5.10-11 par paire de bases à chaque réplication.
Là normalement il y a un-e petit-e malin-e au regard désabusé sur l’école qui lève la main et dit que ça ne joue pas (si on a 3 milliards de bases = 3.10-9 bases /5.10-11 ça ferait de l’ordre de une mutation sur 60 réplications.
on s’informe puisque nous avons a proximité des experts :
« Je ne suis pas sûre qu’il soit possible d’avoir des chiffres beaucoup plus précis. Ce sont des estimations (basées sur les erreurs de la DNA polymérase, sur l’efficacité du système de réparation de l’ADN et sur ????, l’environnement ???) . La seule façon de les vérifier serait de séquencer le génome d’une cellule à chaque génération et de comparer sur plusieurs dizaines de générations….
Mais l’ordre de grandeur reste acceptable (quelques dizaines de nouvelles mutations par génération pour l’homme….) »
Marie-Claude Blatter SIB Institut Suisse de Bioinformatique
Et là on aura de la peine à éviter de discuter comment les méthodes de mesures déterminent le chiffre, ou comment ils ont été calculés … Ce qui peut être très positif.
Ou alors on accepte qu’il n’y a pas de simplement vrai ou faux en sciences et qu’on doit travailler avec des estimations.
savoir travailler avec des incertitudes est une des compétences importantes pour comprendre le monde qui nous entoure, et décider – par exemple s’il faut faire un test génétique.
Le pifomètre, une manière de maîtriser les incertitudes ?
Une publication récente dans Nature (Van Noorden, R., 2015) évoque l’importance de ces calculs approximatifs ( bien sûr c’est d’elle que je me suis inspiré, vous n’imaginez pas que j’invente tout ça ?) doi:10.1038/nature.2015.17140. (Les membres Expériment@l peuvent obtenir ces articles…).
Ils présentent un site : Caladis.org qui permet d’expliciter et de travailler dynamiquement sur les approximations et les degrés d’erreur. Ils proposent de se baser sur BioNumbers

Fig 3: Caladis permet de mesurer dynamiquement les degrés d’incertitude. [img] source Caladis.org
Le nombre de copies d’une protéine dans une cellule ?
Absolute abundance of p53 in cell | Human Homo sapiens | ~160,000 (21,000 to 195,700) | Copies/cell | 100420 | Ma L, Wagner J, Rice JJ…
Milo et al. (2010) Nucl. Acids Res. BNID 100420. BioNumbers
Il y aurait pour la protéine p53 plusieurs dizaines de milliers de copies (entre 21’000 et 195’700) dans une cellule humaine.
Cela donne une idée qui peut compléter les schémas ou l’on ne fait figurer que quelques copies d’une protéine par cellule…
La concentration d’anticorps IgG
La concentration d’anticorps dans le sang est pertinente, car c’est sur la différence de concentration entre les antigènes sur les hématies et les anticorps dans le sérum que repose le modèle (très simpliste et heureusement pas pratiqué dans nos hôpitaux) de « donneur universel ». (Cf notamment : Bio-Tremplins du 28 avril 2007 Tous donneurs universels ?
On trouve bien ceci : Milo et al. (2010) Nucl. Acids Res. BNID 104769 BioNumbers
Concentrations: IgG1= 6.63+-1.17 mg/ml IgG2= 3.22+-1.08 mg/ml IgG3= 0.58+-0.3 mg/ml IgG4= 0.46 mg/ml
Mais on ne trouve pas la concentration d’IgM anti-A ou Anti-B qui permettraient de quantifier la lecture de ce graphique qui donnerait du sens au modèle du donneur universel. Je suis encore à la recherche de ce chiffre si jamais …
Et en physique ?
Ce type de calculs permettant une quantification approximative est aussi appelé problèmes de Fermi ou « back-of-the-envelope calculations« . Les physiciens adorent ! Cf notamment la page Problèmes de Fermi sur Expériment@l qui rassemble plusieurs problèmes que nous vous rappelons.
- D’autres problèmes de Fermi : Octobre
- La muraille de Chine, visible à partir de la lune ?
- Quelle est la hauteur que le jet d’eau peut atteindre
-
Müller, A. (2013). Supersonic Jump. The Physics Teacher, 51(1), 14. DOI:10.1119/1.4772029
- Allart, R. , Cazenave, A. Müller, A. (2015) “Up” – A Fermi question about buoyancy Is it possible for a house to fly by the uplift of balloons, like in the movie “Up?” In Weinstein, Larry. Column Editor, Fermi Questions The Physics Teacher ◆ Vol. 52, 2014 Old Dominion University, Norfolk, VA,USA Intranet.pdf
Le pifomètre, une compétence indispensable au futur biologiste ?
Cette compétence numérique manque souvent dans les formations de biologie. C’est que le fameux rapport Bio2010 – qui a fait référence recommande :
« To successfully undertake careers in research after graduation, students will need scientific knowledge, practice with experimental design, quantitative abilities, and communication skills. « p. 3
« The lack of a quantitative viewpoint in biology courses can result in students who are mathematically talented losing interest in studying the life sciences. While not all students who pursue an education in the bio- medical sciences have an equal interest or predilection for mathematics, it is important that all students understand the growing relevance of quanti- tative science in addressing life-science questions. Thus, a better integra- tion of quantitative applications in biology would not only enhance life science education for all students, but also decrease the chances that mathematically talented students would reject life sciences as too soft. » p. 4
Ils mettent en évidence aussi que donner de la biologie une image trop soft numériquement risque de faire perdre des talents parmi les meilleurs. Ou ils iraient à l’EPFL qui donne l’impression d’être plus numériquement robuste.
Une responsabilité citoyenne?
Le PER désigne la modélisation comme compétence transversale ( pas seulement numérique bien sûr, mais AUSSI numérique) et si nos élèves baissent les yeux gênés devant le premier interlocuteur qui brandit un chiffre en biologie, aurons-nous bien rempli notre mission ? Les aurons-nous rendus capables d’utiliser la biologie pour comprendre et décider dans leur monde ?
S’ils ne comprennent pas en lisant leurs résultats à un test génétique (Angelina Jolie a montré qu’ils se généralisent) qu’un risque 10x plus élevé que la moyenne de la population pour une affection qui se produit une fois sur un million reste négligeable… aurons-nous bien fait notre travail ?
Sources : (Les membres Expériment@l peuvent obtenir ces articles…).
- Allart, R. , Cazenave, A. Müller, A. (2015) “Up” – A Fermi question about buoyancy Is it possible for a house to fly by the uplift of balloons, like in the movie “Up?” In Weinstein, Larry. Column Editor, Fermi Questions The Physics Teacher ◆ Vol. 52, 2014 Old Dominion University, Norfolk, VA,USA Intranet.pdf
- Milo et al. Nucl. Acids Res. (2010) 38: D750-D753. BioNumbers
- NRC. (2003). BIO2010: Transforming Undergraduate Education for Future Research Biologists In Committee on Undergraduate Biology Education to Prepare Research Scientists for the 21st Century (Ed.). Washington, DC.: National Academies Press.
- Müller, A. (2013). Supersonic Jump. The Physics Teacher, 51(1), 14. DOI:10.1119/1.4772029
- Van Noorden, R. (2015). My digital toolbox: back-of-the-envelope biology. Nature. http://doi.org/10.1038/nature.2015.17140
To cite BioNumbers please refer to: Milo et al. Nucl. Acids Res. (2010) 38: D750-D753. When using a specific entry from the database it is highly recommended that you also specify the BioNumbers 6 digit ID, e.g. « BNID 100986, Milo et al 2010 ».
La plateforme Expériment@l vous offre l’accès a ces articles Comment s’inscrire à Expériment@l / Comment Obtenir un article mentionné : Get-a-doi

