Une glissade qui brouille le séquençage
Complément de la page Comment exploiter ces séquences d’ADN en classe ?
Estella Poloni l’AGP, note que « pour 3 séquences ça a visiblement glissé vers 16180. »
Cela signifie qu’une séquence avec une longue suite de C pose problème au séquençage Sanger qui « glisse » et la suite n’est pas fiable.
Pourquoi les CCCCCCCCC posent problème au séquençage ?
Il semblait (Bendall, K. E., & Sykes, B. C., 1995) que le problème découle du fait que certaines personnes ont plusieurs séquences d’ADNmt différentes dans leurs cellules ou mitochondries (hétéroplasmie) au lieu d’un seul type : le séquençage serait flou car l’ADN ne serait pas homogène, il y aurait de fait de nombreuses variantes de longueur légèrement différentes. Actuellement, les raisons n’en sont pas claires, peut-être aussi que la Taq polymérase glisserait à la réplication de l’ADN.
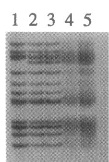
Fig 1 : Gel de 5 fragments d’ADN, piste A seulement. On y voit que les ADN des échantillons 4 et 5 sont « flous » Source : (Bendall, K. E., & Sykes, B. C., 1995)
Le phénomène est souvent observé si on a une variante particulière: C en position 16189.
E. Poloni dit « en effet, par séquençage « classique » (Sanger) et sans clonage, il n’est pas possible de déterminer avec certitude le nombre de répétitions des nucléotides en présence (en particulier le nombre de A après 16182, puis le nombre de C avant 16194, ainsi que la position du T, ou des T quand il y en a plusieurs).
Hétéroplasmie ?
Heteroplasmy is the presence of a mixture of more than one type of an organellar genome (mitochondrial DNA (mtDNA) or plastid DNA) within a cell or individual. It is a factor for the severity of mitochondrial diseases, since every eukaryotic cell contains many hundreds of mitochondria with hundreds of copies of mtDNA, it is possible and indeed very frequent for mutations to affect only some of the copies, while the remaining ones are unaffected. Source : Wikipedia

Fig. 2 : Heteroplasmy 16169 C/T of Nicholas II of Russia. Source : Wikipedia
« A variant commonly found in population screening is a T-to-C transition at nt 16189, resulting in an uninterrupted homopolymeric tract. Direct sequencing of individuals with this variant produces a characteristic blurred sequence in nucleotides beyond the tract. Sequencing clones from these individuals revealed that this is caused by high levels of length heteroplasmy in the homopolymeric tract and low levels of length heteroplasmy in the four adenines following the tract. »
Comment cela influence le séquençage « plus loin ».
Dans cette région entre 16183 et 16193, certains individus ne portent pas de T ; le segment est donc constitué d’un grand nombre de répétitions C, et ceci provoque souvent un glissement lors de la réaction d’amplification. Dans ce cas, le protocole ne permet d’obtenir la séquence entière que dans un sens (par exemple, forward du début jusqu’en 16182, et reverse de la fin jusqu’en 16192) en position 16189.
Du coup un double séquençage forward et reverse depuis l’autre bout du fragment est nécessaire. Il a été effectué pour 3 échantillons. Malgré cela une zone reste difficile à interpréter. Comment on aligne les séquences est présenté dans cette page -> L’alignement : tout un art…
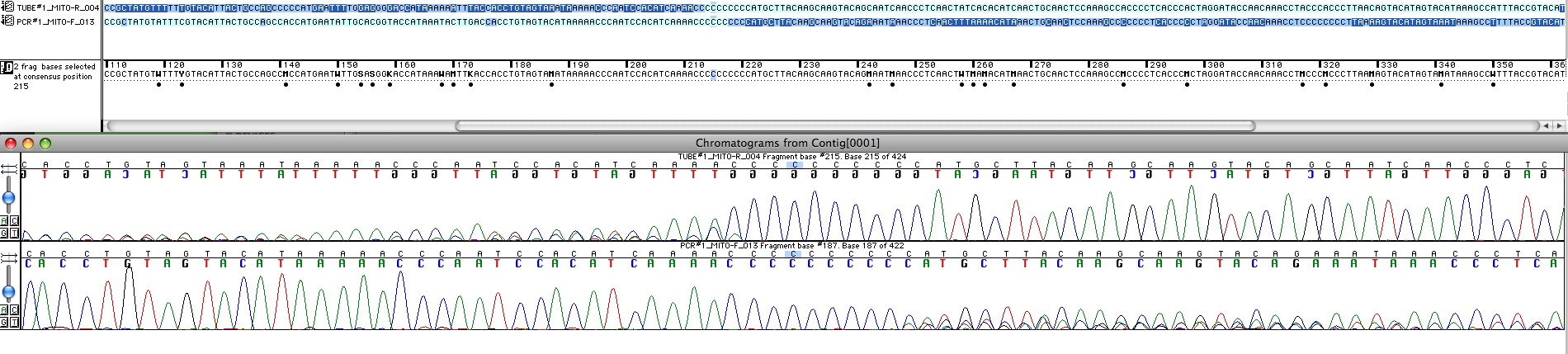
Fig 3 Le séquençage depuis une extrémité "forward" et l'autre "reverse" permettent de déterminer la totalité de la séquence sauf 11 bases au milieu. Cf. L’alignement : tout un art…
On comprend donc pourquoi cette zone (environ 11 pb, des positions 16183 à 16193 inclues) est ignorée pour l’analyse du polymorphisme.
Références
- Bendall, K. E., & Sykes, B. C. (1995). Length heteroplasmy in the first hypervariable segment of the human mtDNA control region. American journal of human genetics, 57(2), 248. pdf
